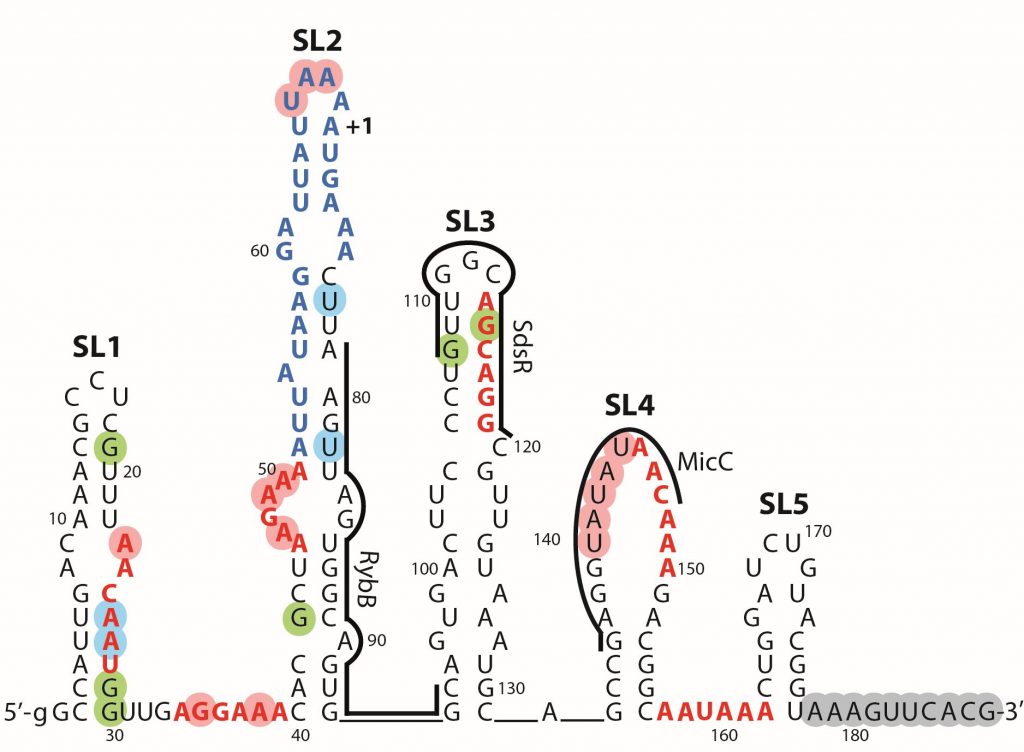
Regulacja translacji przez niekodujące RNA bakterii
Tematem zainteresowań naszego zespołu są mechanizmy molekularne stosowane przez niekodujące RNA bakterii w regulacji translacji. Małe niekodujące RNA, określane też nazwą sRNA (ang. small RNA), uczestniczą w adaptacji bakterii do zmian w środowisku, w tym także biorą udział w regulacji genów wirulencji u bakterii chorobotwórczych. Cząsteczki sRNA mają zwykle około 100 nukleotydów długości i kontrolują translację poprzez wiązanie się z częściowo komplementarnymi sekwencjami w obrębie cząsteczek mRNA. Do efektywnego działania cząsteczki sRNA potrzebują białka opiekuńczego Hfq, które jest homologiczne do białek Sm zaangażowanych w proces splicingu w komórkach eukariotycznych.
Aby wyjaśnić w jaki sposób cząsteczki sRNA oraz białko Hfq uczestniczą w regulacji translacji mRNA badamy oddziaływania pomiędzy nimi stosując głównie metody elektroforetyczne. Analizujemy także struktury cząsteczek RNA i zmiany zachodzące w nich podczas oddziaływań. Rozwijamy także metody oparte o fluorescencyjne i luminescencyjne konstrukty reporterowe do badania przebiegu translacji in vitro, a także metody do monitorowania regulacji translacji w komórkach bakteryjnych.